PRESS RELEASE
2021年3月11日
富士通株式会社
国立大学法人京都大学
富士通と京都大学、AIの推定根拠を説明できる遺伝子変異の病原性推定の検証システムを開発
ゲノム医療における未知の遺伝子変異に基づく治療検討を支援
富士通株式会社(注1)(以下、富士通)と国立大学法人京都大学(注2)(以下、京都大学)大学院医学研究科の奥野恭史教授らの研究グループは、国立研究開発法人日本医療研究開発機構(以下、日本医療研究開発機構)の「臨床ゲノム情報統合データベース整備事業」(注3)において、特定の遺伝子変異(注4)が何らかの疾患の原因になる可能性(以下、病原性)の有無を推定し、その推定根拠を添えて治療方針を検討する医師やゲノム医療の研究者に示すことができるAIを活用した検証システム「MGeND Intelligence」(エムジェンド インテリジェンス)を開発しました。京都大学は、2021年4月より、共同研究者や協力機関などに対して「MGeND Intelligence」の利用提供を予定しています。
「MGeND Intelligence」は、遺伝子変異の病原性の有無を高精度に推定できる病原性推定AI、推定の根拠を示す説明可能AI、関連する論文記載の検索を支援する文献探索支援AIから構成されます。京都大学が2018年に公開した臨床ゲノム情報統合データベース「MGeND」と連携し、病原性の有無が未知の変異も含めて医療従事者や研究者による遺伝子変異の調査・臨床的解釈の作業を支援します。
「MGeND Intelligence」を利用することで、がんをはじめとしたゲノム医療での治療方針検討など、診療支援につながるとともに、患者にとって最適な医療提供が加速されることが期待できます。
背景
ゲノム医療では、患者の個々の遺伝子変異に対して病原性の有無を知ることが重要です。患者の遺伝子に病原性のある変異を見つけることができれば、その変異に対して治療法を検討することができます。しかし、膨大な遺伝子変異のうち疾患への関連性が明らかになっている変異はごく一部にすぎません。病原性の有無が未知の遺伝子変異からは、疾患の治療に役立つ情報を得ることができていないことが課題となっています。
富士通と京都大学は、2016年11月より日本医療研究開発機構が推進する「臨床ゲノム情報統合データベース整備事業」に参画し、医療従事者や研究者による遺伝子変異の検討作業をAIで支援するための研究開発に取り組んできました。そしてこのたび、未知の遺伝子変異に対して病原性の有無を推定し、その推定根拠を説明することができるAIを活用した検証システムを開発し、京都大学より共同研究者や協力機関などへ公開します。
「MGeND Intelligence」の特長
「MGeND Intelligence」は、富士通と京都大学が共同で開発した、遺伝子変異の検討作業の支援を目的とした検証システムです。株式会社富士通研究所(注5)(以下、富士通研究所)が保有する3つの技術である、複雑な事象も表現できるグラフ構造(注6)のデータを学習する機械学習技術、AIの推定根拠を示す説明可能なAI技術、大量文献から変異や疾患などの関係を高精度に抽出できる自然言語処理技術の応用により実現されたものです。
患者の遺伝子変異情報を「MGeND Intelligence」に入力すると、機械学習技術を活用した病原性推定AIにより病原性有無を推定し、説明可能AIにより推定結果とともに推定の根拠の説明文を生成して表示します。また、変異に関連する公共データベースの情報やAIによる推定の確からしさを、周辺変異の情報とともに多角的に可視化して表示することや、自然言語処理技術を活用した文献探索支援AIにより、対象変異に関連する論文を探し記載箇所を特定して表示することができるため、医療従事者や研究者が自ら推定結果の根拠を収集することも可能です。
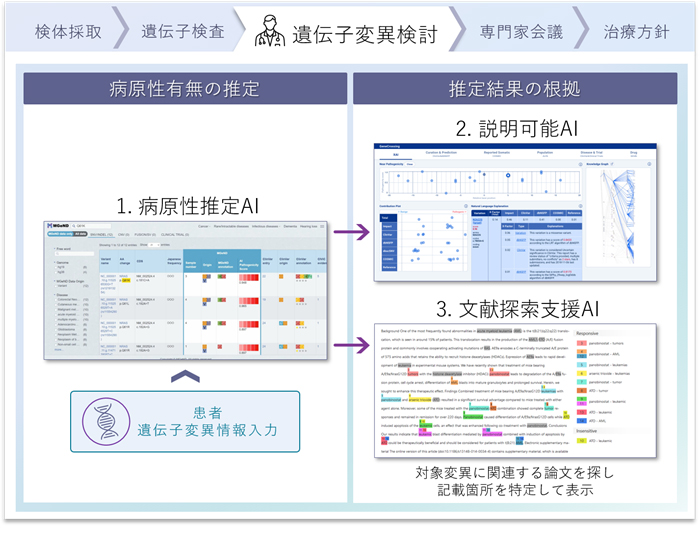 検証システム構成図
検証システム構成図-
医療従事者や研究者が理解しやすい推定根拠を自然文で説明
「Deep Tensor」を用いた推定では、ナレッジグラフ上で推定に大きく寄与したグラフノード(以下、推定因子)を示すことができるという特徴(注9)があります。しかし、病原性の判定は複数の根拠をもとに総合的に行われるため、ナレッジグラフ上で推定根拠となった部分を示すだけでは病原性推定の説明として十分ではありませんでした。そこで今回、どのような説明をすればAIによる病原性推定の結果を信頼できるかについて、奥野教授らのグループおよびゲノム医療の研究者へヒアリングを行い、複数の説明パターンを抽出しました。その上で、「Deep Tensor」で得られる推定因子から各説明パターンの適合する度合いを計算し、その度合いの高い複数のパターンをもとに推定根拠を説明する文章を生成します。医療従事者や研究者は推定根拠の説明文を一読するだけで、推定に大きく寄与した重要な根拠を把握することが可能です。 -
病原性を判断する根拠となる論文の調査を支援
大量の論文から対象とする変異と任意疾患との関係を記述している部分を検索し、論文中の該当部分をハイライトして表示します。関連情報を調べるために、例えば、大量の文献から変異と薬剤との関係性を記述した文章だけを明示的に表示することも可能です。これにより、過去論文に含まれている推定根拠を容易に調べることができ、既知の事実に基づく推定結果の検討時間を大幅に短縮できます(注10)。
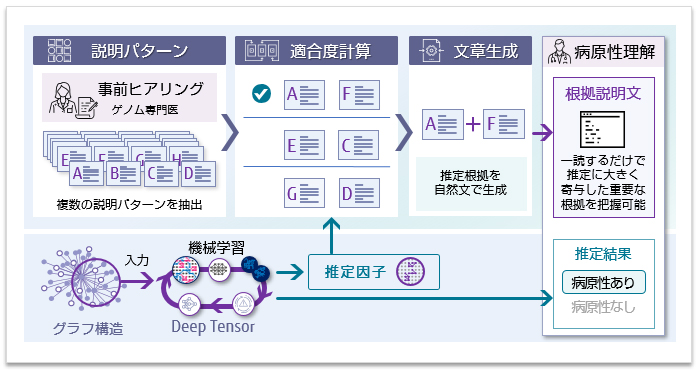 推定根拠の説明文生成のフロー図
推定根拠の説明文生成のフロー図東京大学医科学研究所(注11)ヒトゲノム解析センターの協力のもと、遺伝性疾患治療を題材に、未知変異(注12)について病原性推定から根拠獲得まで、病原性推定システム全体の評価試験を行い、臨床面の観点からの示唆をシステムに反映させました。この結果、いずれの変異に対しても病原性の推定結果は妥当であり、根拠を説明する文章も専門医から見て適切なものとなったことを確認しました。
例えば、今回検証したある変異(注13)では、評価試験で用いたシステム構築時には病原性が未知で、その後、病原性ありと判定された変異ですが、病原性推定AIは「病原性あり」と正しく推定しました。また、評価者は、AIが生成した説明文を読むことでAIによる推定根拠の概要を把握した上で、病原性ありと考えるのに十分な根拠を集めることができました。
今後の展開
富士通では、今回の成果をもとに説明機能のさらなる充実を図り、電子カルテを主とした病院情報システムにおけるゲノム医療を支援する機能を新規開発し、全国の中核拠点及び拠点病院、連携病院への導入を目指します。
京都大学は、当該日本医療研究開発機構事業の共同研究者や協力機関などを中心に、「MGeND Intelligence」の利用提供を予定しています。
商標について
記載されている製品名などの固有名詞は、各社の商標または登録商標です。
注釈
-
注1富士通株式会社:
本社 東京都港区、代表取締役社長 時田 隆仁。 -
注2国立大学法人京都大学:
所在地 京都府京都市、総長 湊 長博。 -
注3臨床ゲノム情報統合データベース整備事業:
政府のゲノム医療実現推進協議会 中間とりまとめを踏まえ、ゲノム情報と疾患特異性や臨床特性などの関連について日本人を対象とした検証を行い、臨床および研究に活用することができる臨床情報と遺伝情報を統合的に扱うデータベースを整備するとともに、その研究基盤を利活用した先端研究開発を一体的に推進する事業。 -
注4遺伝子変異:
遺伝子の多様性のこと。ここでは便宜上、集団内に一定以上の割合で存在する疾患に関係しない遺伝子多型を含め、「遺伝子変異」という表現を用いる。 -
注5株式会社富士通研究所:
本社 神奈川県川崎市、代表取締役社長 原 裕貴。 -
注6グラフ構造:
ノード(点)をエッジ(線)で接続した形状として表現される情報で、代表的な知識表現形式のひとつ。 -
注7ナレッジグラフ:
論文や研究成果といった形で表現される情報を集め、互いに関連するもの同士を接続したグラフ構造の集合体で計算機で扱う知識を格納するために使われる。 -
注8Deep Tensor:
人やモノのつながりを表現できるグラフ構造のデータに対して高精度な解析を可能とする富士通独自の機械学習技術。 -
注9ナレッジグラフ上で推定に大きく寄与したグラフノード(推定因子)を示すことができるという特徴:
推定に寄与したグラフノードを示すことができるという技術については下記プレスリリースを参照。
「AIの推定理由や根拠を説明する技術を開発」(2017年9月20日) -
注10既知の事実に基づく推定結果の検討時間を大幅に短縮できます:
検討時間を短縮した事例については下記プレスリリースを参照。
「新しいAIによるがんゲノム医療の効率化を東大医科研との共同研究で実現」(2019年11月6日) -
注11東京大学医科学研究所:
所在地 東京都港区、所長 山梨 裕司。 -
注12未知変異:
評価に用いたシステムに搭載されているAI推定のモデル構築時点で病原性の有無が不明だった遺伝子変異で、現在は病原性の有無が判明しているもの。 -
注13今回検証したある変異:
遺伝子MYO7Aの変異:R1873Q。
本件に関するお問い合わせ
富士通株式会社
-
富士通コンタクトライン(総合窓口)
0120-933-200(通話無料)受付時間: 9時~17時30分(土曜日・日曜日・祝日・富士通指定の休業日を除く)
-
Webでのお問い合わせ
入力フォームへ当社はセキュリティ保護の観点からSSL技術を使用しております。
京都大学大学院
プレスリリースに記載された製品の価格、仕様、サービス内容などは発表日現在のものです。その後予告なしに変更されることがあります。あらかじめご了承ください。
